与脊髓灰质炎、SARS/MERS、流感、埃博拉、寨卡等其他RNA病毒类似,新冠病毒的转录复制过程由自身编码的依赖RNA的RNA聚合酶(RNA-dependent RNA polymerase,简称RdRP)来主导完成。有些RdRP如脊髓灰质炎病毒的3Dpol蛋白可相对独立地实现RNA合成,而冠状病毒的RdRP蛋白nsp12在发挥转录复制功能时还需要nsp7和nsp8这两个辅助因子参与,工作机制较为复杂。当前备受关注的核苷类抗新冠药物瑞德西韦(remdesivir)、法匹拉韦(favipiravir)均以其三磷酸核苷效应分子的形式直接靶向nsp12,但相关作用机制尚不明晰。因此,从结构生物学和酶学水平探索新冠病毒RdRP-nsp7-nsp8转录复制机器的工作机制和核苷类药物的作用机制,对了解新冠病毒的本质特征和研发有效药物具有重要意义。
2020年5月22日,上海科技大学和中国科学院武汉病毒研究所合作,结合双方在结构病毒学和RdRP研究中的长期积累,在Cell杂志上发表题为“Structural basis for RNA replication by the SARS-CoV-2 polymerase”(《新冠病毒聚合酶实现RNA复制的结构基础》)的文章,报道了新冠病毒聚合酶在催化RNA合成过程中的“转位前”和“转位后”两种重要状态的构象特征(图1及图2)。合作团队通过筛选十余种RNA成功获得了经历四轮催化反应的“转位后”复合物结构(PDB号7BZF,分辨率3.26埃,图2A),进而通过改变复合物下游模板序列以允许瑞德西韦效应分子参与催化而获得了一种“转位前”复合物结构,其中瑞德西韦成功 “混入”了产物RNA链中,并且完成了向上游的转位(PDB号7C2K,分辨率2.93埃,图2B)。研究团队通过生化活性数据与结构结果相支撑,首次验证了瑞德西韦在“混入”产物链后与双链RNA退出通道中的关键氨基酸S861产生位阻效应,从而导致产物链在瑞德西韦首次“混入”后需再经历三轮催化方发生链终止(也称“i+3延迟终止”)的机制(图3及图4)。本研究首次精细描绘出了新冠病毒“RdRP-nsp7-nsp8”转录复制机器处在工作状态下的核心特征,展现了复合体辅助部件参与反应的分子机制和结构基础,并为瑞德西韦和法匹拉韦等候选药物的效应分子如何精确靶向抑制病毒RNA合成进而发挥药效活性提出了机制解释。
上海科技大学王权助理教授和武汉病毒所吴继芹助理研究员等五人为本文共同第一作者。清华大学教授、中科院生物物理所研究员,上科大兼职教授饶子和院士、中科院武汉病毒所龚鹏研究员、上科大王权助理教授为本文共同通讯作者。本研究得到国家重点研发计划、国家自然科学基金等项目的支持。
论文链接:https://www.cell.com/cell/fulltext/S0092-8674(20)30629-2
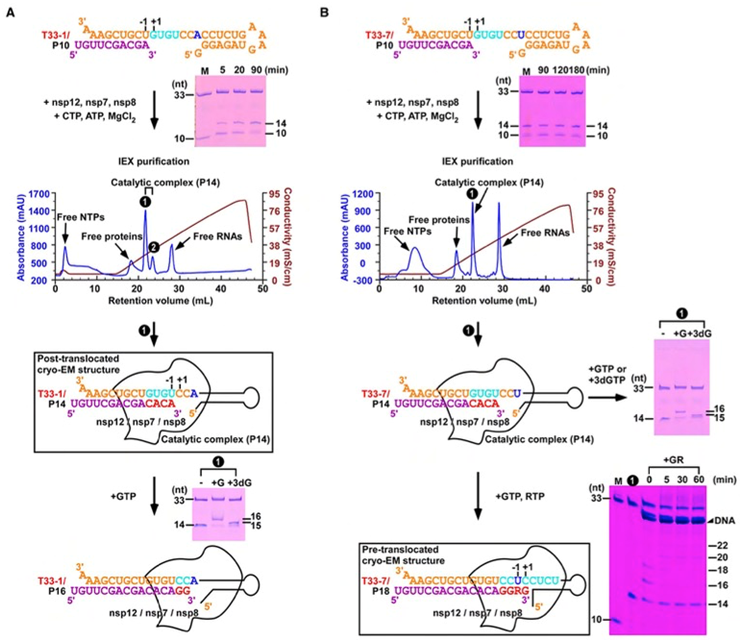
图1. 新冠病毒聚合酶催化复合物的组装。A)T33-1/P10 RNA经历四轮催化后形成T33-1/P14复合物,可通过色谱纯化后用于冷冻电镜测试。B)T33-7/P10 RNA在形成T33-7/P14复合物并经色谱纯化后,可继续经历四轮催化得到混入瑞德西韦(R)的复合物。
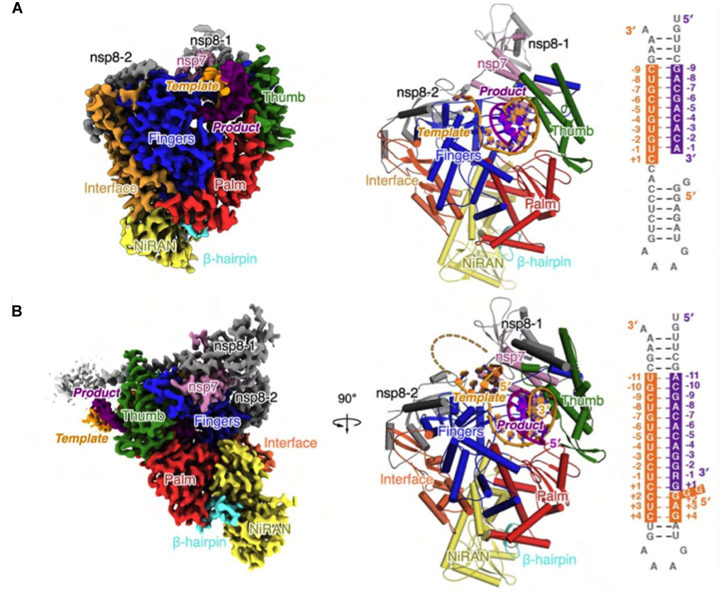
图2. 新冠病毒聚合酶催化复合物结构。A)T33-1/P14“转位后”复合物。B)“混入”了瑞德西韦的T33-7/P18 “转位前”复合物。

图3. 聚合酶复合物结构提示瑞德西韦等核苷类分子的抑制机制。A)在 “转位前”复合物结构中,瑞德西韦“混入”后发生了一次转位,到达“-1”位;而新一轮的催化反应已发生,但处于“转位前”状态。B)K545和S682在NTP识别中发挥关键作用。C)结构分析提示法匹拉韦效应分子可较好地“模拟”GTP与模板咆嘧啶碱基(C)配对。D)在 “转位前”复合物结构中,瑞德西韦和尿嘧啶碱基(U)在“-1”位的相互作用与A:U碱基对相似。E)结构分析提示,当瑞德西韦向上游移动到“-4”位时,将与S861的侧链产生位阻效应。
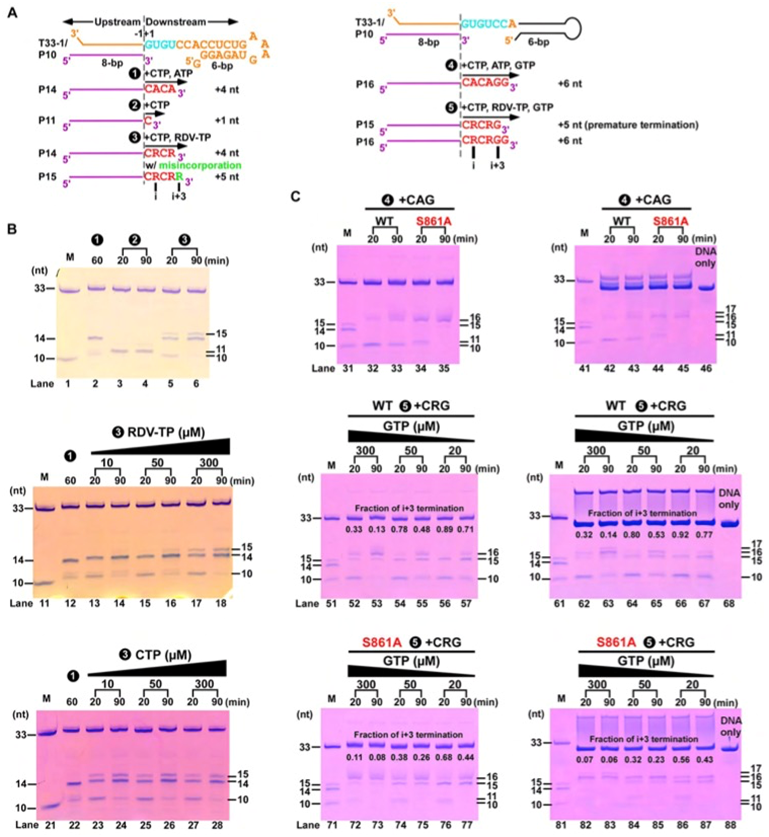
图4. S861位阻效应的酶学验证。A)基于T33-1/P10 RNA的催化反应流程图。B)瑞德西韦效应分子(RDV-TP)具有不弱于ATP的反应性且易诱导错配反应的发生。C)上:当CTP/ATP/GTP作为底物时,主要产物为P16;中/下:当瑞德西韦效应分子替代ATP时,出现了与延迟终止机制相关的P15产物,且S861A突变体生成的P15产物少于野生型,从而证实了延迟终止和S861与瑞德西韦之间的位阻效应有关。
 附件下载:
附件下载:
科学研究